Capitolo 2 Trattamento di dati T.A.C.
2.1 Tomografia assiale computerizzata
La tomografia computerizzata è una tecnica radiologica relativamente recente destinata ad esaminare le strutture interne del corpo a livello macroscopico senza effetto oscuramento dovuto al sovrapporsi delle strutture, rendendo superfluo l’uso di mezzi di contrasto. Contrariamente alla radiologia tradizionale, di cui si riporta un esempio in fig. 1‑1, è in grado di risolvere le piccole differenze di contrasto tra strutture vicine di densità simile (es. parti molli).
Il principio si basa sulla ricostruzione dell'immagine di sezioni corporee di spessore prefissato a partire da una serie di misure di assorbimento di fotoni, provenienti da un fascio "a pennello" od "a ventaglio" lungo numerose traiettorie di attraversamento della sezione considerata. Utilizzando infatti fotoni X di energia sufficientemente elevata e privilegiando, quindi, le interazioni del tipo effetto Compton, l’attenuazione fotonica dipende per lo più dalla densità fisica del tessuto attraversato; si può pertanto stabilire una proporzionalità tra il coefficiente lineare di attenuazione e la densità fisica in ogni punto. Da questi coefficienti, opportunamente rielaborati (sottraendo a ciascuno il valore corrispondente all’acqua, moltiplicando quest’ultimo per il risultato ottenuto e ancora per 1000), si giunge ad attribuire a ciascun punto di ogni tessuto attraversato dai fotoni X, un valore di attenuazione espresso in Hounsfield (H), dal nome dell’inventore della TAC .
Le caratteristiche principali sono rappresentate da:
- elevata possibilità di contrasto, che può accrescersi con l’incremento della dose somministrata, soprattutto tra osso e tessuti molli;
- basso contrasto tra tessuti molli;
- ricchezza di informazioni morfologiche;
- possibilità di somministrazione di mezzo di contrasto.
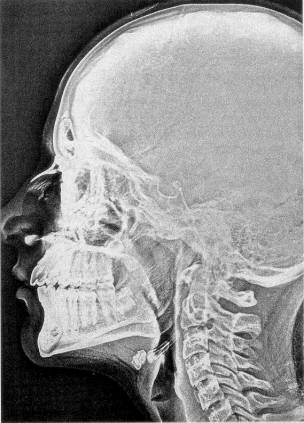
Figura 1‑1 Immagine radiografica tradizionale
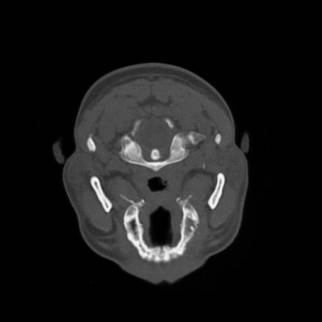
Figura 1‑2 Immagine TAC relativa ad una sezione trasversale del cranio.
In figura 1‑3 viene presentato un esempio delle moderne apparecchiature diagnostiche. L’interazione tra computers e macchina diagnostica permette un miglior controllo della macchina stessa e l’elaborazione immediata dei dati acquisiti.

Figura 1‑3 Moderne apparecchiature diagnostiche
2.2 Digitalizzazione
É il procedimento di trasformazione delle immagini originali in forma digitale.
Nelle moderne apparecchiature mediche ciò è automatico (nella digitalizzazione automatica la conversione analogico-digitale viene effettuata dalla macchina stessa) per cui si pone unicamente il problema di trasferire i dati attraverso reti di comunicazione o per mezzo dei diversi mezzi di supporto per l’archiviazione, effettuando le eventuali conversioni di formato.
Tale operazione è specifica per ogni apparecchiatura; infatti la realizzazione e, soprattutto, la diffusione di formati standard è di recente istituzione. Nella maggior parte dei casi i costruttori (come ad esempio General Electrics e Siemens) hanno impiegato formati proprietari, diversi a volte non solo da marchio a marchio, ma anche da modello a modello, nell'ambito di ciascuna metodica. L'esigenza di diversi algoritmi specifici per la traduzione di ogni formato-dati comporta un notevole aumento di costi e tempi oltre che un difficile aggiornamento; inoltre attualmente l’industria non fornisce ragguagli sulla maggior parte dei formati proprietari, che risultano pertanto non accessibili. Vi sono quindi vincoli rilevanti nella possibilità di interfacciare liberamente le stazioni grafiche.
Lo sviluppo attuale dell'informatica medica percorre per questo motivo la strada della creazione di formati standard (es. ACR/NEMA DICOM 3) e dello sviluppo di reti per la trasmissione dei dati sempre più affidabili, veloci, ed a basso costo, in virtù, anche, di programmi di compressione più potenti.
Lo standard ACR/NEMA è stato proposto nel 1985 da American College of Radilogists (ACR) e National Electric Manufacturers Association (NEMA). Per infromazioni specifiche si consiglia di consultare ACR/NEMA Standards Pubblications.
Il DICOM 3 (Digital Imaging and Communication in Medicine) è a tutti gli effetti un protocollo di rete che fornisce specifiche per i diversi livelli di comunicazione. Prevede le facilities degli standard di rete per l’interconnessione (TCP-IP e ISO-OSI), la negoziazione dei messaggi, la specificazione object-oriented di set di dati e classi di servizi.
I formati più diffusi prevedono una dimensione di 512x512 pixel, memorizzati per linee, a 12 bit (4096 tonalità di grigio). Da notare che il numero dei livelli di grigio, pur essendo sufficientemente significativo, è fondamentalmente determinato dalla difficoltà di realizzare convertitori analogico/digitale veloci per un numero di bit superiore, perciò negli anni a venire si assisterà forse a un ampliamento della scala.
In figura 1‑4
viene riportato un formato ACR-NEMA, che oltre ai valori dei pixel memorizzati
per righe prevede un header ASCII, strutturato a etichette (tag) con campi di lunghezza variabile,
contenente informazioni sul paziente, sulle modalità di acquisizione, ecc.
(0000x8,0000x0) UL IdentifyingGroupLength VR=<UL> VL=<0x4> [0x194]
(0000x8,0000x1) UL LengthToEnd VR=<UL>
VL=<0x4> [0x80538]
(0000x8,0000x8) CS ImageType VR=<CS>
VL=<0x16>
<ORIGINAL\PRIMARY\AXIAL>
(0000x8,000x16) UI SOPClassUID
VR=<UI> VL=<0x1a>
<1.2.840.10008.5.1.4.1.1.2>
(0000x8,000x18) UI SOPInstanceUID VR=<UI> VL=<0x30>
<1.2.840.113619.2.1.587.2776917735.2.21.867345919>
(0000x8,000x20) DA StudyDate VR=<DA> VL=<0x8>
<19970626>
(0000x8,000x21) DA SeriesDate VR=<DA> VL=<0x8>
<19970626>
(0000x8,000x22) DA AcquisitionDate VR=<DA> VL=<0x8>
<20041222>
(0000x8,000x23) DA ImageDate VR=<DA> VL=<0x8>
<19970626>
(0000x8,000x30) TM StudyTime VR=<TM>
VL=<0x6> <172051>
(0000x8,000x31) TM SeriesTime VR=<TM>
VL=<0x6> <172329>
(0000x8,000x32) TM AcquisitionTime VR=<TM>
VL=<0x6> <203010>
(0000x8,000x33) TM ImageTime VR=<TM>
VL=<0x6> <172519>
(0000x8,000x50) SH AccessionNumber VR=<SH>
VL=<0x0> <>
(0000x8,000x60) CS Modality VR=<CS>
VL=<0x2> <CT>
(0000x8,000x70) LO Manufacturer VR=<LO>
VL=<0x12> <GE MEDICAL
SYSTEMS>
(0000x8,000x80) LO InstitutionName VR=<LO> VL=<0x1c>
<Istituto Ortopedico Rizzoli>
(0000x8,000x90) PN ReferringPhysicianName VR=<PN>
VL=<0x4> <AMB.>
(0000x8,0x1010) SH StationName VR=<SH>
VL=<0x8> <CT01_OC0>
(0000x8,0x1030) LO StudyDescription VR=<LO> VL=<0x12>
<MASSICCIO FACCIALE>
(0000x8,0x103e) LO SeriesDescription VR=<LO> VL=<0xa>
<MASSICCIO>
(000x10,0000x0) UL PatientGroupLength VR=<UL>
VL=<0x4> [0x60]
(000x10,000x10) PN PatientName VR=<PN> VL=<0xe> <RESTA
ANTONIO>
(000x10,000x20) LO PatientID VR=<LO> VL=<0xc> <MASS.
FACC.>
(000x10,000x30) DA PatientBirthDate VR=<DA> VL=<0x0> <>
(000x10,000x40) CS PatientSex VR=<CS>
VL=<0x2> <M>
(000x10,0x1010) AS PatientAge VR=<AS> VL=<0x4> <034Y>
(000x10,0x1030) DS PatientWeight VR=<DS>
VL=<0x8> <0.000000>
(000x10,0x21b0) LT AdditionalPatientHistory VR=<LT>
VL=<0x0> <>
(000x18,0000x0) UL AcquisitionGroupLength VR=<UL>
VL=<0x4> [0x10a]
(000x18,000x10) LO ContrastBolusAgent VR=<LO> VL=<0x4>
<NONE>
(000x18,000x22) CS ScanOptions VR=<CS>
VL=<0xc> <HELICAL
MODE>
(000x18,000x50) DS SliceThickness VR=<DS>
VL=<0x8> <3.000000>
(000x18,000x60) DS KVP VR=<DS>
VL=<0x4> <120>
(000x18,000x90) DS DataCollectionDiameter VR=<DS> VL=<0xa> <250.000000>
(000x18,0x1020) LO SoftwareVersion VR=<LO> VL=<0x2>
<05>
(000x18,0x1100) DS ReconstructionDiameter VR=<DS>
VL=<0xe>
<230.0000000000>
(000x18,0x1110) DS DistanceSourceToDetector VR=<DS> VL=<0x10> <1099.3100585938>
(000x18,0x1111) DS DistanceSourceToPatient VR=<DS>
VL=<0xa>
<630.000000>
(000x18,0x1120) DS GantryDetectorTilt VR=<DS>
VL=<0xa>
<-16.500000>
(000x18,0x1130) DS TableHeight VR=<DS>
VL=<0xa>
<193.399994>
(000x18,0x1150) IS ExposureTime VR=<IS>
VL=<0x4> <1000>
(000x18,0x1151) IS XrayTubeCurrent VR=<IS>
VL=<0x4> <180>
(000x18,0x1160) SH FilterType VR=<SH>
VL=<0xc> <BODY
FILTER>
(000x18,0x1190) DS FocalSpot VR=<DS>
VL=<0x2> <1>
(000x18,0x1210) SH ConvolutionKernel VR=<SH>
VL=<0x4> <BONE>
(000x18,0x5100) CS PatientPosition VR=<CS>
VL=<0x4> <HFS>
(000x20,0000x0) UL ImageGroupLength VR=<UL>
VL=<0x4> [0x164]
(000x20,0000xd) UI StudyInstanceUID VR=<UI> VL=<0x2c> <1.2.840.113619.2.1.587.3616229583.867345651>
(000x20,0000xe) UI SeriesInstanceUID VR=<UI>VL=<0x2e>
<1.2.840.113619.2.1.587.3599904764.2.867345809>
(000x20,000x10) SH StudyID VR=<SH>
VL=<0x4> <587>
(000x20,000x11) IS SeriesNumber VR=<IS>
VL=<0x2> <2>
(000x20,000x12) IS AcquisitionNumber VR=<IS>
VL=<0x2> <1>
(000x20,000x13) IS ImageNumber VR=<IS>
VL=<0x2> <21>
(000x20,000x32) DS ImagePositionPatient VR=<DS>
VL=<0x22>
<-115.000000\-99.164268\-43.773796>
(000x20,000x37)
DS ImageOrientationPatient VR=<DS> VL=<0x36>
<1.000000\0.000000\0.000000\0.000000\0.958820\0.284015>
(000x20,000x52) UI
FrameOfReferenceUID VR=<UI>
VL=<0x38>
<1.2.840.113619.2.1.587.3599904764.2.867345809.3227013939>
(000x20,000x60) CS Laterality VR=<CS>
VL=<0x0> <>
(000x20,0x1040) LO PositionReferenceIndicator VR=<LO>
VL=<0x2> <OM>
(000x20,0x1041) DS SliceLocation VR=<DS>
VL=<0xe>
<-14.4012212753>
(000x28,0000x0) UL ImagePresentationGroupLength
VR=<UL>
VL=<0x4> [0x9e]
(000x28,0000x2) US SamplesPerPixel VR=<US>
VL=<0x2> [0x1]
(000x28,0000x4) CS PhotometricInterpretation VR=<CS>
VL=<0xc>
<MONOCHROME2>
(000x28,000x10) US Rows VR=<US>
VL=<0x2> [0x200]
(000x28,000x11) US Columns VR=<US>
VL=<0x2> [0x200]
(000x28,000x30) DS PixelSpacing VR=<DS> VL=<0x1a> <0.4492187500\0.4492187500>
(000x28,00x100) US BitsAllocated VR=<US>
VL=<0x2> [0x10]
(000x28,00x101) US BitsStored VR=<US>
VL=<0x2> [0x10]
(000x28,00x102) US HighBit VR=<US>
VL=<0x2> [0xf]
(000x28,00x103) US PixelRepresentation VR=<US>
VL=<0x2> [0x1]
(000x28,00x120) XS PixelPaddingValue VR=<XS>
VL=<0x2> [0xf830]
(000x28,0x1052) DS RescaleIntercept VR=<DS>
VL=<0x6> <-1024>
(000x28,0x1053) DS RescaleSlope VR=<DS>
VL=<0x2> <1>
(0x7fe0,0000x0) UL PixelDataGroupLength VR=<UL>
VL=<0x4> [0x80008]
(0x7fe0,000x10) OX PixelData VR=<OW> VL=<0x80000>
Figura 1‑4 Header ACR-NEMA
2.3 Ricostruzione tridimensionale
La ricostruzione 3D da sequenze di immagini bidimensionali TAC costituisce un procedimento attualmente consolidato per la cui realizzazione sono disponibili in commercio svariati programmi.
Prima di descrivere in dettaglio le singole procedure, onde evitare equivoci interpretativi al lettore, definiamo il significato che verrà attribuito ad alcuni termini ricorrenti.
Nell'ambito della visualizzazione scientifica per oggetto si intende la parte o le parti anatomiche che si desidera visualizzare o riprodurre (un singolo organo, un apparato, un distretto, l’intero corpo). Esso è dotato di una o più superfici e di un contenuto che definiamo struttura (parenchimale, ossea, liquida ecc.). Quando è rappresentato in sezione, la sua superficie diviene una linea bidimensionale chiusa, il profilo, che può in realtà essere composto da più linee distinte, come avviene nel caso in cui l'oggetto possegga cavità interne.
Tutti i programmi si basano su due tecniche di ricostruzione: per superfici o per volumi[1].
La ricostruzione per superfici, mostrata in figura 1‑5, considera solamente i dati relativi alla superficie dell’oggetto, ignorandone la struttura. Una volta definito il profilo di interesse per ogni sezione, i dati residui possono essere eliminati. L'oggetto può quindi essere rappresentato come una sequenza di curve approssimanti (dette spline) impilate nello spazio, connesse da un mosaico di poligoni (usualmente triangoli) o intercettate da una superficie complessa.
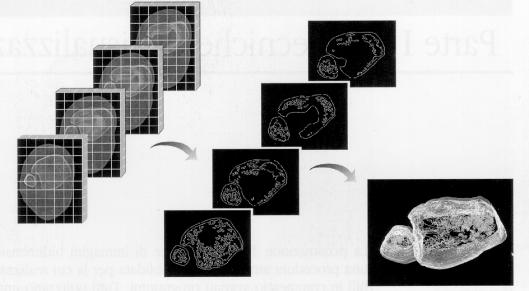
Figura 1‑5 Ricostruzione per superfici
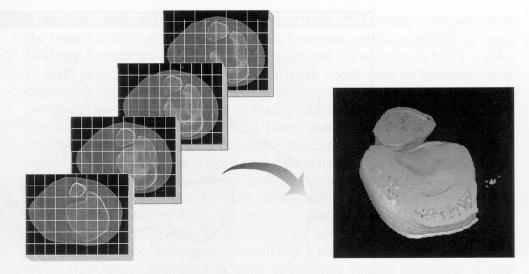
Figura 1‑6 Ricostruzione volumetrica
La tecnica di ricostruzione volumetrica, mostrata in figura 1‑6, invece ricostruisce spazialmente sia la superficie sia la struttura dell'oggetto, impiegando una matrice 3D di voxel[2]. Se dal punto di vista della rappresentazione grafica il risultato può essere analogo con entrambe le tecniche, ben diversi sono il numero di informazioni contenute nella ricostruzione 3D dell'oggetto - la sola superficie nel primo caso, superficie e struttura nel secondo - e, di conseguenza, le possibilità di ulteriore elaborazione.
La caratteristica saliente della rappresentazione volumetrica, molto sfruttata in campo ingegneristico, è in realtà la possibilità di lavorare sugli oggetti intendendoli realmente come solidi, quindi dotati di massa, peso, caratteristiche meccaniche e fisiche, ecc. (modeling). Attualmente questa caratteristica non viene utilizzata appieno nelle applicazioni biomediche, dal momento che dell'oggetto si tende a riprodurre solamente l’aspetto esteriore, o eventualmente la struttura interna. Tuttavia, sono già in fase di sviluppo modelli ricostruiti elettronicamente non solo per una valutazione visiva ma anche per simulazioni fisiche o biomeccaniche.
Si passa ora alla descrizione delle singole procedure che intervengono nel processo di ricostruzione.
Registrazione
Qualora le immagini presentino problemi di allineamento, dovuti a movimenti del paziente o a tecniche di digitalizzazione manuali, si rende necessario un passo preliminare di image registration per il quale sono oggi disponibile programmi automatici o semiautomatici.
Questo procedimento si avvale prevalentemente di algoritmi di tipo otptical flow (features tracking) o mutua informazione [MBR].
Filtraggio
Le applicazioni dei filtri hanno principalmente l’obbiettivo di:
- sfumare;
- evidenziare.
L’immagine ottenuta dovrà possedere le stesse caratteristiche di quella originale, cioè possedere lo stesso set di voxel ma con densità differenti.
Normalmente i filtri vengono realizzati applicando all’immagine una qualche funzione di natura algebrica, differenziale o integrale.
Il numero di quelli disponibili è veramente molto elevato. Tra i più diffusi si annoverano quelli per:
· eliminare determinate frequenze d’intensità (passa-basso, passa-alto, passa-banda);
· smussare le irregolarità (smooth);
· evidenziare i contorni (edge enhancement);
· variare il contrasto o l’intensità.
La figura 1‑7 mostra un filtraggio per il rilevamento e l’evidenziazione dei contorni.
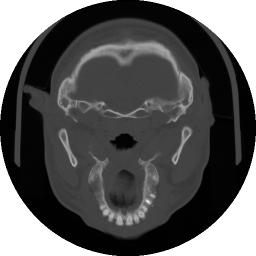
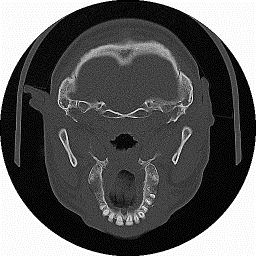
Figura 1‑7 Filtraggio di tipo find-edges enhanced
Interpolazione
L'interpolazione si può intendere come quella operazione di ricampionamento di una immagine su una diversa griglia di riferimento. Spesso le immagini che originano dalle moderne procedure non sono uniformemente campionate, cioè presentano differenze tra i singoli assi generando voxel non cubici.
È un problema che si verifica in particolar modo nella ricostruzione da sequenze di immagini 2D nella direzione ortogonale al piano delle immagini, nella quale il passo di campionamento è spesso superiore a quello delle immagini stesse e talvolta non uniforme.
Gli algoritmi d’interpolazione più diffusi sono di tipo lineare, sinc, e spline.
Segmentazione
La segmentazione è il processo che suddivide un'immagine nelle sue parti componenti, poi usate a scopo di analisi, descrizione e riconoscimento a più alto livello.
Si possono comunemente suddividere le tecniche di segmentazione in due categorie:
1. tecniche di estrazione di regioni (ossa, parti molli, ecc.):
· sogliatura;
· region growing;
2. tecniche di estrazione di contorni:
·
marching
cubes;
·
modelli
deformabili:
·
snakes;
·
baloon;
La sogliatura è una operazione di selezione in base ai toni di grigio. è estremamente rudimentale e come tale di semplice implementazione inoltre non prevede la possibilità di considerare un qualche criterio di connettività.
Il region growing è una tecnica che prende in considerazione le caratteristiche dell’immagine per raggruppare i voxel e formare regioni secondo un criterio di omogeneità. Partendo da alcune regioni iniziali (fase di suddivisione), queste vengono fuse (fase di fusione) per costituire regioni più estese, fino a quando il procedimento sia possibile.
Il marching cubes è una tecnica per la costruzione di iso-superfici 3D.
L’algoritmo originale è stato proposto da Lorensen e Cline [LOR] e prevede due passi:
1. si individua la superficie corrispondente a un determinato valore e si creano i triangoli.
2. per assicurare la qualità dell’immagine si calcolano le normali alla superficie nei vertici di ogni triangolo, in modo da poter applicare ad esempio un Gouraud shading.
La strategia complessiva è di tipo dividi et impera. Dapprima si localizza la superficie in un cubo logico di otto voxel, presi a gruppi di quattro da due slices adiacenti, poi si determina come la superficie interseca il cubo e poi si passa al successivo. Dato che ci sono otto vertici per ogni cubo e due stati (interno ed esterno) esistono 256 possibilità che vengono codificate in un’apposita tavola. In realtà i casi base sono 14 e i restanti costituiscono permutazioni ottenibili per simmetria e rotazione. Si arriva a una triangolazione all’interno di ciascun cubo e si applica un’interpolazione lineare per trovare le intersezioni della superficie sugli spigoli.
Le snakes sono state introdotte da Kass [KAS] per modellare e segmentare oggetti in immagini 2D. Appartengono a una classe di modelli deformabili in cui si cerca di minimizzare una funzione energia associata.
Il successo della segmentazione attraverso le snakes è limitato dalla sensibilità dell’algoritmo nei confronti del rumore e delle condizioni iniziali: ciò è dovuto principalmente alle ottimizzazioni locali che spesso impediscono di trovare il minimo globale dell’energia, e all’approssimazione poligonale discreta per i contorni.
Il balloon consiste in una geometria, ad esempio l’approssimazione poligonale di una sfera, da deformare fino a coincidere con la superficie di un oggetto. Il comportamento desiderato del modello è determinato dal costo locale dina funzione associata a ciascun vertice e determinata dalla somma di tre termini: un potenziale di deformazione che espande i vertici verso la superficie dell’oggetto, un termine immagine che identifica proprietà come i contorni e si oppone all’espansione, e un termine che mantiene la topologia del modello costringendo ogni vertice a rimanere al centro dei suoi vicini. Per approfondimenti si consiglia la lettura di [MIL] in cui questo procedimento è applicato a un volume 3D di dati TAC.
Estrazione della regione d’interesse
Può accadere che il processo automatico di segmentazione difetti e possa essere necessario l’ausilio dell’operatore, che indichi la regione di interesse.
A questo livello non è più possibile intervenire in modo automatico, bensì attraverso strumenti software, che implicano una componente di manualità più o meno accentuata. Evidentemente l’operazione è caratterizzata da una certa discrezionalità per cui l’esito dipende fortemente dall’esperienza dell’operatore.
In figura 1‑8 si mostra il caso frequente di un paziente sottoposto a TAC , la cui immagini contiene anomalie dovute alla presenza di amalgami dentali.
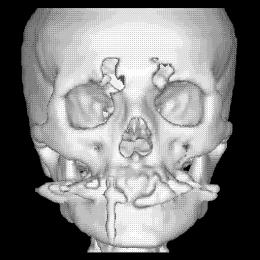
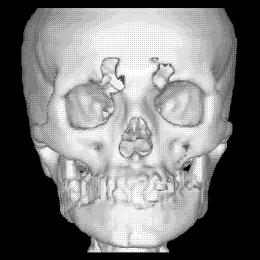
Figura 1‑8 Immagine originale e regione d’interesse
2.4 Visualizzazione
È la rappresentazione sullo schermo del computer della immagine tridimensionale, attraverso le note modalità di computer graphics, con le possibilità di effettuare ingrandimenti e rimpicciolimenti (zoom), traslazioni e rotazioni.
Lo scopo è rendere l’immagine in modo tale che possa essere percepita nella sua tridimensionalità.
Le tecniche di visualizzazione tridimensionale sono state studiate a fondo nel campo delle applicazioni mediche e il loro impiego come ausilio clinico è ormai consolidato. Maggiori informazioni si possono trovare in [BIA].
In genere si ricorre ai seguenti metodi di visualizzazione:
·
slice
imaging;
·
surface
rendering;
· volume rendering;
e tra questi quello sicuramente più sfruttato è il surface rendering.
Slice imaging
Lo scopo di questa operazione è di suddividere l’oggetto in sezioni altrimenti non disponibili (ortogonali e oblique), visualizzabili come immagini a livelli di grigio.
Le operazioni di estrazione e di interpolazione implicano calcoli per la regolarizzazione delle caratteristiche dei voxel. Nonostante la facilità di implementazione e la velocità di visualizzazione, questo metodo non è in grado di offrire una percezione complessiva della tridimensionalità.
Surface Rendering
É una tecnica di visualizzazione per superfici.
L’illusione di tridimensionalità viene creata evidenziando il limite tridimensionale della struttura (contouring) e inserendo alcune sorgenti luminose fittizie.
Quest’ultima metodica, denominata shading, ombreggiatura, origina dalla considerazione secondo la quale nei meccanismi naturali della visione un oggetto, per essere visibile, deve essere adeguatamente illuminato; vi saranno parti dell'oggetto che risulteranno nascoste, perché non riflettenti, e parti meno evidenti, perché poco riflettenti.
I gruppi di fattori che determinano l’intensità di illuminazione sono :
· le condizioni di illuminazione e le coordinate del punto di vista;
· le proprietà ottiche dell'oggetto, (il colore, la trasparenza od opacità, le sue caratteristiche di superficie liscia, rugosa ecc.).
Se l’oggetto è opaco basta considerarne le proprietà di superficie; se invece non lo è, bisognerà considerarne complessivamente le caratteristiche ottiche, fatto quest'ultimo che nella visione naturale si reperisce molto più raramente.
Bisogna, d'altra parte, considerare che una struttura anatomica può essere, e spesso è, disomogenea dal punto di vista delle sue proprietà ottiche.
Per i nostri scopi si tratterà di definire un modello ottico adeguato della superficie dell'oggetto.
Ciascuno dei parametri, qui di seguito citati, deve essere preso in considerazione per ottenere un'immagine corretta:
· distanza;
· riflessione diffusa;
· riflessione speculare;
· colore della luce riflessa;
· trasparenza;
· visione nello spazio.
Da questa premessa di base nascono le tecniche di scene-space shading, in cui la superficie rappresenta l’insieme dei punti nei quali il gradiente di cambio di densità è massimo, di object-space shading dato da una valutazione ed interpolazione geometrica sulla superficie stessa, e di view-space shading, che prende in considerazione solo le parti della superficie proiettate nello spazio di proiezione.
La figura 1‑9 mostra un esempio di surface rendering. Si noti come grazie all’ombreggiatura si avverte la sensazione di tridimensionalità.
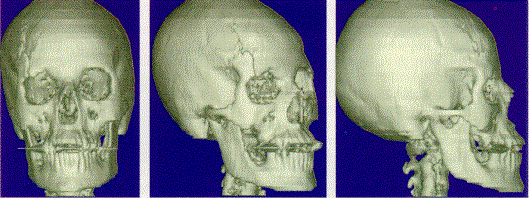
Figura 1‑9 Ricostruzione 3D in surface rendering
Volume rendering
É una tecnica di visualizzazione di volumi dove la rappresentazione tridimensionale avviene attribuendo a ciascun voxel proprietà di opacità e di colore dipendenti dai tessuti, tenendo conto delle proprietà ottiche dell’intero oggetto (fondamentalmente transmittanza e riflettanza alle interfacce tra tessuti diversi).
Questo presuppone a monte (in genere all’atto della segmentazione), un lavoro per assegnare a ogni voxel un valore di opacità e di colore. Qualora in uno stesso voxel siano rappresentati più tessuti, si procederà a un calcolo integrale.
Altra modalità di segmentazione nel volume rendering è data dalla rappresentazione di sole superfici di interfaccia tra tessuti diversi anziché di interi blocchi. Il rendering in questo caso si prefigge di determinare il colore da assegnare a ogni pixel nelle singole proiezioni attraverso la determinazione dell’insieme di voxel che contribuiscono alla formazione del pixel stesso, come avviene nei processi di voxel projection e ray casting.
Nel primo caso per ogni data proiezione si può determinare l’ordine delle sezioni, delle singole file di voxel di ciascuna di esse ed infine di ogni singolo voxel nell’ambito di ciascuna fila.
Nel caso di ray casting invece i voxel che influenzano le proprietà di ciascun pixel vengono identificati mandando un raggio di luce fittizia dal pixel alla scena; i voxel coinvolti nelle determinazioni del pixel in questione saranno quelli intercettati dal raggio.
Per la rappresentazione di volumi binari si possono usare tecniche di memorizzazione per riga e per sezione dei voxel caricati (es tecniche di rectangular array); si può altresì dividere lo spazio in regioni secondo una rappresentazione gerarchica (octrees). Così se una regione contiene voxel a densità nulla la suddivisione si ferma; in caso contrario essa prosegue fino ad altre otto sottoregioni e così via finché possibile. Questa tecnica ben si adatta, per ogni rotazione della scena, a ordinare i voxel dal più lontano al più vicino.
Il grey volumes rendering prevede che tutti i voxel abbiano lo stesso colore, ma le loro opacità sono determinate basandosi sulle varizioni di intensità simulando una riproiezione radiografica. Si può altresì attribuire una scala di colori fittizi differenziali a ciascun pixel secondo la luce riflessa (una variante del ray casting).
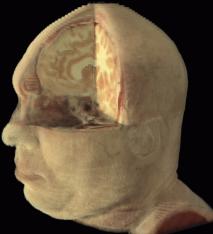
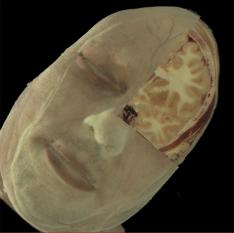
Figura 1‑10 : Esempio di volume rendering
Surface versus volume rendering
Nessuna delle due metodiche prese in esame è aliena dal creare falsi positivi o negativi, ma questi dipendono dalla sequenza delle metodiche e delle loro applicazioni.
Il surface rendering non è la sola tecnica che trascuri alcuni dettagli del cranio e della superficie encefalica; piuttosto bisogna ammettere che la geometria definisce una superficie che non può contenere strutture fini o comunque troppo dettagliate, qualunque sia la definizione delle tecniche di acquisizione in esame.
Il volume rendering ha il vantaggio di garantire un rapido colpo d'occhio dei contenuti, specialmente nel caso in cui le strutture di interesse siano diffuse (es. tumori infiltranti), mentre la forza del surface rendering consiste proprio nella proprietà di garantire operazioni interattive dipendenti dalla possibilità di orientare diversamente le superfici in esame .
2.5 Altre applicazioni della Registrazione
Nel paragrafo precedente si è citata la registrazione di immagini quale mezzo per risolvere i problemi di allineamento delle slices, tale tecnica ha un campo di applicazione più vasto e infatti le procedure per la ricostruzione dei tessuti molli esposte nel capitolo 5 possono essere viste come particolari casi di registrazione.
Volendo dare una definizione più generale di registrazione possiamo descriverla come l’applicazione di una trasformazione ad una entita’ tale che alcune delle sue proprieta’ diventino congruenti con quelle di una entita’ di riferimento.
Le principali applicazioni nell’ambito bio-medico riguardano:
· la registrazione di dati di uno stesso paziente acquisite mediante modalità diverse [Multi Modality Image Registration], sono generalmente trasformazioni di tipo rigido. Nel corso della tesi si è sperimentato un applicativo di questo tipo che è [MIRIT].
· La registrazione di dati relativi a pazienti diversi oppure di dati aquisiti in età diverse di uno stesso paziente.
· Registrazione funzionale alla segmentazione, prevede la registrazione con un atlante, cioe’ un insieme di dati piu’ esteso e standardizzato che puo’ contenere informazioni riguardo la funzionalità o la topologia dei vari organi
Come esposto in [MBR] è conveniente pensare la registrazione come il moto di un insieme di dati verso l’altro, in questo modo possiamo classificare le varie tecniche in base a due fattori: il tipo di moto che determina quali trasformazioni sono permesse e il potenziale d’attrazione che indica da quali forze il moto è guidato.
Tipi di moto:
· Rigido. è composto da una rotazione seguita da una traslazione e può quindi essere espresso mediante 6 parametri. L’oggetto trasportato conserva la sua forma durante il moto.
· Elastico. Nel corso del moto l’oggetto viene assimilato ad un solido elastico. Il numero di parametri necessari a descrivere tale trasformazione può avere l’ordine di grandezza delle centinaia.
· Fluido. Assimilando l’oggetto ad un corpo fluido le trasformazioni si complicano ulteriormente con conseguente aumento dei parametri
· Libero. Espresso mediante trasformazioni che agiscono localmente su ciascuna delle parti dell’oggetto trasportato: non é previsto nessun criterio per controllare l’omogeneità delle trasformazioni applicate.
Potenziale di attrazione:
· corrispondenza tra punti
· Intrinseci: punti caratteristici che è possibile individuare automaticamente.
· Estrinseci: punti caratteristici posizionati manualmente sul paziente oppure nei dati relativi. Sono esempi di questo tipo di registrazione le procedure presentate nei paragrafi 3 e 6 del capitolo 5.
· Corrispondenza tra curve o superfici:
· Momenti ed assi principali:
· Similitudine tra i Voxel: E’ un esempio di questo tipo di registrazione la procedure presentata nel paragrafo 5 del capitolo 5.